R: 支持向量机(Support Vector Machine,简称SVM)
在数据科学和机器学习领域中,支持向量机(Support Vector Machine,简称SVM)是一种强大的监督学习算法,常用于分类和回归分析。它的优点之一是可以适用于复杂的数据集,并且在高维空间中表现良好。在本文中,我们将使用R语言和一些R自带的数据集来介绍如何使用支持向量机进行建模和模型评价。
1. 准备数据
首先,让我们选择一个适合的数据集。在R中,有许多自带的数据集可供使用。我们选择一个相对复杂的数据集,以便更好地展示支持向量机的应用。
我们选择的数据集是iris,它包含了鸢尾花的测量数据。这个数据集有四个特征:花萼长度、花萼宽度、花瓣长度和花瓣宽度,并且有三种不同的鸢尾花品种:Setosa、Versicolor和Virginica。
# 加载iris数据集
data(iris)2. 划分数据集
在开始建模之前,我们需要将数据集划分为训练集和测试集。训练集用于训练模型,测试集用于评估模型的性能。
# 设置随机种子,确保结果可重复
set.seed(123)
# 划分数据集为训练集和测试集
train_index <- sample(1:nrow(iris), 0.7 * nrow(iris))
train_data <- iris[train_index, ]
test_data <- iris[-train_index, ]3. 建立模型
接下来,我们使用训练集建立支持向量机模型。在这里,我们将使用e1071包中的svm()函数来实现。
# 加载e1071包
library(e1071)
# 使用svm()函数建立支持向量机模型
svm_model <- svm(Species ~ ., data = train_data, kernel = "radial")4. 模型优化
支持向量机有许多参数可以调整,例如核函数类型、惩罚参数等。为了使模型更准确,我们可以使用交叉验证来选择最佳的参数组合。
# 使用交叉验证选择最佳的参数组合
tune_result <- tune(svm, Species ~ ., data = train_data, kernel = "radial",
ranges = list(cost = c(0.1, 1, 10), gamma = c(0.1, 1, 10)))
best_model <- tune_result$best.model5. 模型评价
最后,我们使用测试集评估模型的性能。我们将使用混淆矩阵、准确率和其他指标来评价模型的表现。
# 预测测试集
predicted <- predict(best_model, test_data)
# 计算混淆矩阵
confusion_matrix <- table(predicted, test_data$Species)
print(confusion_matrix)##
## predicted setosa versicolor virginica
## setosa 14 0 0
## versicolor 0 17 0
## virginica 0 1 13# 计算准确率
accuracy <- sum(diag(confusion_matrix)) / sum(confusion_matrix)
print(paste("准确率:", accuracy))## [1] "准确率: 0.977777777777778"SVM的可视化
让我们继续使用经典的鸢尾花(iris)数据集来演示支持向量机(SVM)的结果可视化。
# 加载必要的包
library(e1071)
library(ggplot2)
# 加载iris数据集
data(iris)
# 创建训练集和测试集
set.seed(123)
train_index <- sample(1:nrow(iris), 0.7 * nrow(iris))
train_data <- iris[train_index, ]
test_data <- iris[-train_index, ]
# 使用svm函数建立模型
svm_model <- svm(Species ~ ., data = train_data, kernel = "radial")
# 预测类别
test_data$predicted <- predict(svm_model, test_data)
# 可视化结果
ggplot(test_data, aes(x = Petal.Length, y = Petal.Width, color = predicted, shape = Species)) +
geom_point(size = 3) +
scale_color_manual(values = c("#FF0000", "#00FF00", "#0000FF")) +
scale_shape_manual(values = c(16, 17, 18)) +
labs(title = "SVM Result Visualization on Iris Dataset", color = "Predicted Species", shape = "True Species")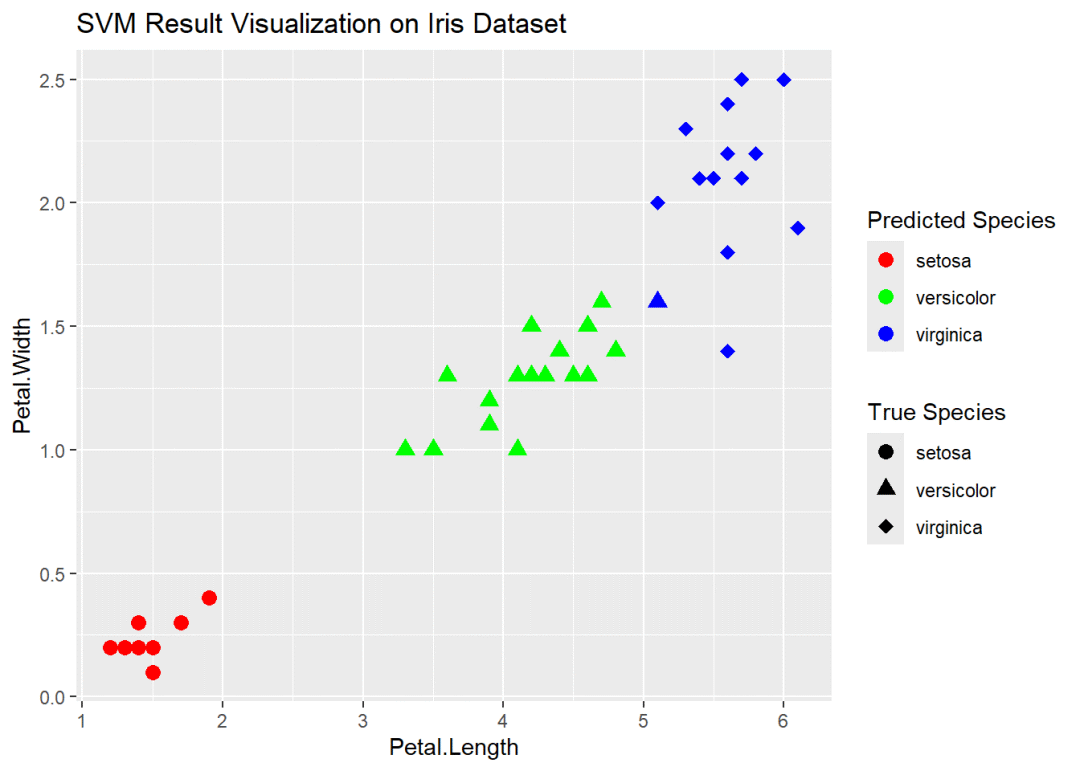
这样的可视化可以帮助我们直观地了解SVM模型在iris数据集上的分类效果。
原文地址:https://blog.csdn.net/weixin_46587777/article/details/137574840
免责声明:本站文章内容转载自网络资源,如本站内容侵犯了原著者的合法权益,可联系本站删除。更多内容请关注自学内容网(zxcms.com)!
