scanMiR:使用R语言预测 miRNA 结合位点
生信碱移
scanMiR 结合预测
scanMiR,一款R语言工具包,能够高效地扫描任何自定义序列上的典型和非典型miRNA结合位点,估计解离常数并预测聚合的转录物抑制。
最近,几项高通量研究试图阐明miRNA–mRNA靶向的生化决定因素。在一项发表于Science的研究中,McGeary等人使用纯化的Ago–miRNA复合体结合RNA结合-n-序列(RBNS)来确定六种miRNA(miR-1、let-7a、miR-7、miR-124、miR-155和lsy-6,基于它们的保守序列和现有功能数据选择)对任何可能的12个核苷酸长(12-mer)序列的亲和力。他们展示了不同典型位点类型的亲和力和相应解离常数(KD)与细胞环境中观察到的抑制非常吻合,这大大优于重新训练的TargetScan7版本。为了将这些发现推广到其他miRNA,作者使用实证获得的数据训练了一个卷积神经网络(CNN)。他们证明了基于通过CNN获得的亲和值的抑制分数在另一种细胞类型中的12种不同miRNA上比TargetScan7提高了约50%。

▲ McGeary等人的研究
尽管如此,虽然McGeary等人(2019)提供的模型和实验数据在目标预测方面代表了重大进展,但它们并没有提供给更广泛的用户群。事实上,基于生化模型的占用分数已被添加到TargetScan8,但这样广泛使用的平台未能提供所需的灵活性:例如,TargetScan只包括少数几种物种,仅涵盖转录组的一部分(每个蛋白质编码基因的单个策划UTR),不提供非典型或开放阅读框结合位点的信息,且对自定义序列的应用非常繁琐。最后,尽管R和Bioconductor在生物信息学中很普遍,但基于R的miRNA-靶标预测工具相对稀缺。
因此,来自瑞士的研究人员试图通过在Bioconductor框架内整合和扩展McGeary的生化模型,提供一个通用的、灵活的、用户友好的软件包和伴随的Web应用程序scanMiR。scanMiR于2022年发表于「Bioinformatics」杂志,能够高效地扫描任何自定义序列上的典型和非典型miRNA结合位点,估计解离常数并预测聚合的转录物抑制。作者还提供了一个灵活的3′-补充对齐,允许系统地预测可能的结合位点以及导致RNA剪切的位点。此外,scanMiR提供了几个新的可视化功能,以便于miRNA-靶标候选选择。scanMiR预测的mRNA抑制与测量的mRNA折叠变化比TargetScan分数显著更相关。最后,作者还将scanMiR集成到了一个web应用程序,可供大部分人群使用。
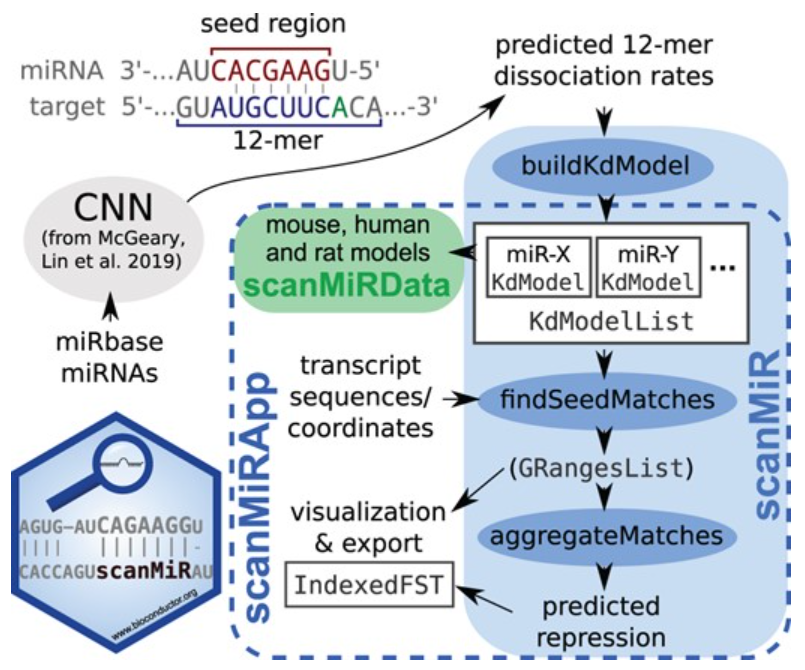
▲ scanMiR的整体设计
一、miRNA的介绍
microRNA(miRNA)是一类小型、单链的非编码RNA分子,包含21至23个核苷酸。在植物、动物和一些病毒中发现的miRNA,参与RNA沉默和基因表达的转录后调控。miRNA与mRNA分子中的互补序列配对,通过以下一个或多个过程使该mRNA分子沉默:
-
切割mRNA链为两部分。
-
通过缩短其poly(A)尾部使mRNA稳定性降低。
-
翻译mRNA为蛋白质。
miRNA与RNA干扰(RNAi)途径中的小干扰RNA(siRNA)相似,不同之处在于miRNA源自RNA转录本中折叠回自身形成的短发夹结构,而siRNA源自更长的双链RNA区域。人类基因组可能编码超过1900种miRNA,尽管最近的分析表明这个数字接近于2300。然而,在手动策划的miRNA基因数据库MirGeneDB中,只有大约500种人类miRNA被确认为真正的miRNA。成熟的miRNA是活跃的RNA诱导的沉默复合体(RISC)的一部分,该复合体包含Dicer和许多相关蛋白。RISC也被称为microRNA核糖核蛋白复合体(miRNP);含有miRNA的RISC有时被称为“miRISC”。
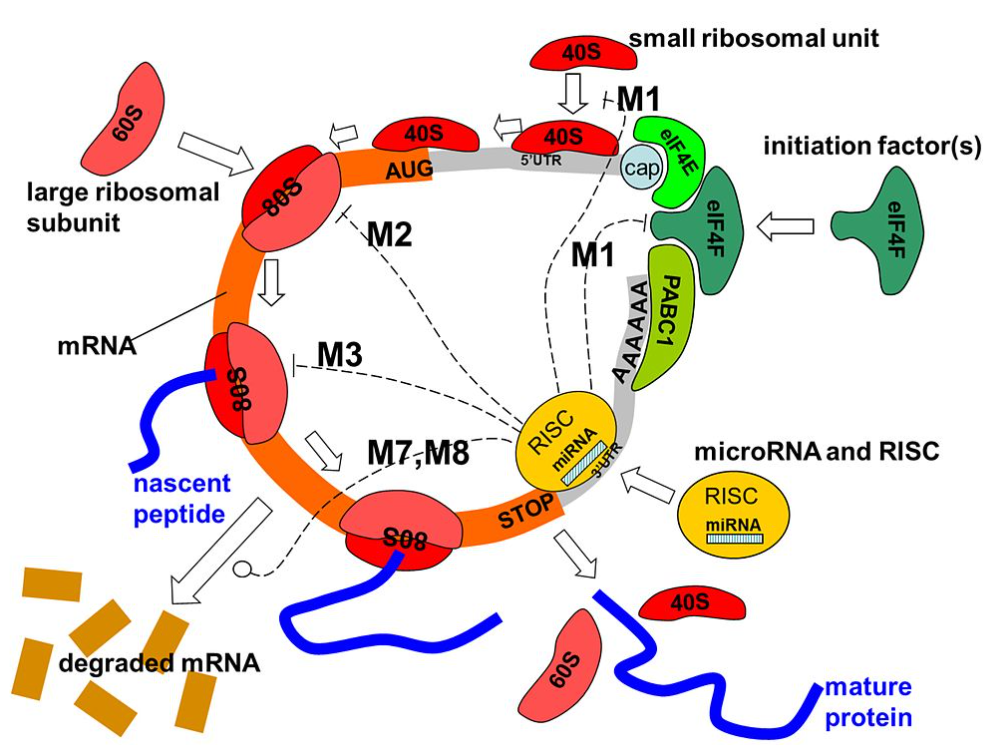
▲ 上图展示了几种翻译抑制机制:M1)对起始过程的干扰,阻止起始复合体的组装或招募40S核糖体亚基;M2)对核糖体组装的干扰;M3)对翻译过程的干扰;M7、M8)对mRNA的降解。40S和60S是核糖体的轻型和重型组分,80S是结合到mRNA的组装核糖体,eIF4F是翻译起始因子,PABC1是Poly-A结合蛋白,而“cap”是mRNA圆化所需的mRNA帽结构(可以是正常的m7G帽或修饰的A帽)。mRNA的起始可以通过招募40S到位于5'UTR区域的内部核糖体进入位点(IRES)以独立于帽的方式进行。RNA沉默的实际工作由RISC执行,其中主要的催化亚单位是阿尔戈蛋白(AGO)之一,而miRNA充当识别特定mRNA序列的模板。
阿尔戈蛋白(Ago)家族的成员是RISC功能的核心。阿尔戈蛋白对miRNA诱导的沉默是必需的,包含两个保守的RNA结合域:PAZ域可绑定成熟miRNA的单链3'端,PIWI域在结构上类似于核糖核酸酶-H,并用于与引导链的5'端相互作用。它们绑定成熟miRNA,并将其定位于与目标mRNA的相互作用。一些阿尔戈蛋白,例如人类Ago2,可以直接切割靶转录本;阿尔戈蛋白还可能招募额外的蛋白来实现翻译抑制。
miRNA在许多哺乳动物细胞类型中丰富存在,也存在于细胞外循环miRNA中。细胞外循环miRNA被释放到包括血液和脑脊液在内的体液中,有可能作为多种疾病的生物标志物。此外,许多miRNA在进化上是保守的,这意味着它们具有重要的生物学功能。
二、R包安装
devtools::install_github("omarwagih/ggseqlogo")
BiocManager::install("ETHZ-INS/scanMiR")
三、简单的示例
详情查看代码注释哈:
library(BiocStyle)
library(scanMiR)
# miRNA的序列,可以从miRbase数据库获得
miRNA <- "UUAAUGCUAAUCGUGAUAGGGGUU"
# 加载示例mRNA序列数据
data("SampleTranscript")
print(SampleTranscript)
#[1] "CAACGGATTGGGGGCAGCATTCTCGTCGTTTCGGATCCGGCCTTCTCTTAATAGTGATCTCCGATTACTCCATTGGCGGTCCTGCGAAGTTGATATTCATCAGAAGTTTGTTCTGTGCAACATTGTAGATTTTCATGGCACTCTTCTTCGCCGGCGCATGATCACCCACATTATGCAGATTGCGTCACACATCTTCCTTCTTCGGGTCGGCGTATTAAATTTAGCAAGGTTCCAGTGGTTGAGCACCTAAGTAATTGCACGTCGAACATCTAACTGCTAATAACTATGCTCGGCTAATAACGCCCCAGTCCCAAACAGGGAAGCTTAACTTTGGCATCAGCGTGGTCCAGTGATCGATACAGACGAATGCGGGCTAAGTCAGCATGAATGCTGGTCCTTTTGGCCTAGTACGTTCAGTTTTATCATTTTGGGCGGTAGGCACATTGGCAATTCGGGAGCAATATAGAGTAACGACCCCTATCACGTCCGCAGCATTAAATGGACACTAAACACTCGCGAATCGTATCGGATTGCCTTCTATTCGCGTATCGCGTCGTCAGAGTAAGAGCTAGAGTGTGATAATTGACTGACCCAATTGAAGGGATATATACGGGAAGCGGTTGACCGGAAGCACAGGTTATAGACCCTGCTGCTTGGCTCTCTACCCCCTTCCTTGTCGGAGAGCATTTTGAGCATTATGATGCAAGGGAATCTTCGCTCGGGGGTCGCCGATCCACGCTATCGCAAGATGGTTCAGGCTTCGTTCTCCGAAGAGACACATTAGTTATACCTGCTTTTGAGCGCGCAATCCCAAGGGACCATCGACACACGGACCGGGTCACACGACACACCGGACAGCAAACAACGTCCAAGTGACCTTCAGATTTCAGCAGACTAG"
# 执行匹配
matches <- findSeedMatches(SampleTranscript, miRNA, verbose = FALSE)
matches # 简单看一下返回的结果
#GRanges object with 2 ranges and 3 metadata columns:
# seqnames ranges strand | type p3.score note
# <Rle> <IRanges> <Rle> | <factor> <integer> <Rle>
# [1] seq1 491-498 * | 8mer 12 TDMD?
# [2] seq1 692-699 * | 7mer-m8 0 -
# -------
# seqinfo: 1 sequence from an unspecified genome; no seqlengths
# 简单看看结合模式
viewTargetAlignment(matches[1], miRNA, SampleTranscript)
#miRNA 3'-UUGGGGAUAGUGC-UAA-UCGUAAUU-5'
# |||||||||||| ||||||||
#target 5'-...NGAGUAACGACCCCUAUCACGUCCGCAGCAUUAAAU...-3'
# 当然,也可以输出为图片
viewTargetAlignment(matches[1], miRNA, SampleTranscript, outputType = "plot")
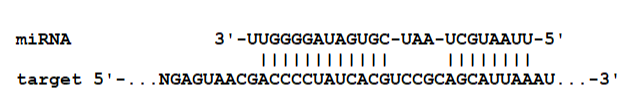
更多函数:
-
aggregateMatches: aggregateMatches -
assignKdType: assignKdType -
c-method: miRNA affinity models -
conservation: conservation -
dummyKdData: Create dummy log_kd per 12-mer data -
findSeedMatches: Predicting and characterizing miRNA binding sites -
get3pAlignment: Finds 3' complementary binding of a miRNA -
get8merRange: get8merRange -
getKdModel: getKdModel -
getKmers: getKmers -
getMatchTypes: getMatchTypes -
getRandomSeq: getRandomSeq -
getSeed8mers: getSeed8mers -
KdModel: miRNA affinity models -
KdModel-class: miRNA affinity models -
KdModel-methods: miRNA affinity models -
KdModelList: KdModelList -
KdModelList-class: KdModelList -
KdModelList-methods: Methods for the 'KdModelList' classes -
KdModelList-methods-method: Methods for the 'KdModelList' classes -
plotKdModel: plotKdModel -
removeOverlappingRanges: removeOverlappingRanges -
SampleKdModel: Example KdModel (hsa-miR-155-5p) -
SampleTranscript: Example transcript sequence -
show-method: miRNA affinity models -
summary-method: miRNA affinity models -
summary-method: Methods for the 'KdModelList' classes -
viewTargetAlignment: viewTargetAlignment
参考链接:
-
https://academic.oup.com/bioinformatics/article/38/9/2466/6533438?login=false
-
McGeary S.E. et al. (2019) The biochemical basis of microRNA targeting efficacy. Science, 366, eaav1741.
-
Wiki
简单分享到这
各位早点休息
原文地址:https://blog.csdn.net/weixin_49214410/article/details/142788107
免责声明:本站文章内容转载自网络资源,如本站内容侵犯了原著者的合法权益,可联系本站删除。更多内容请关注自学内容网(zxcms.com)!
