提交高通量测序原始数据到 SRA --- 操作流程
❝写在前面
由于最近在提交课题数据到 NCBI 数据库,整理了相关笔记。本着自己学习、分享他人的态度,分享学习笔记,希望能对大家有所帮助。推荐先按顺序阅读往期内容:
1. 提交高通量测序数据到 GEO --- 说明书
目录
-
1 注册 NCBI 账号 -
2 准备要上传的原始数据 -
3 填写数据信息 -
3.1 填写提交者信息 -
3.2 填写一般信息 -
3.3 填写项目信息 -
3.4 填写样本类型 -
3.5 填写样本属性 -
3.6 填写 SRA Metadata -
3.7 上传文件 -
3.8 检查并提交
-
在发表文章之前往往需要将高通量测序的数据上传到 NCBI 数据库。上传的数据可以分为两类:① Raw data(fastq文件)通常上传到 SRA、② Processed data(counts matrix、RDS 等)通常上传到 GEO。本文详细介绍提交高通量测序 raw data 到 SRA 的操作流程。
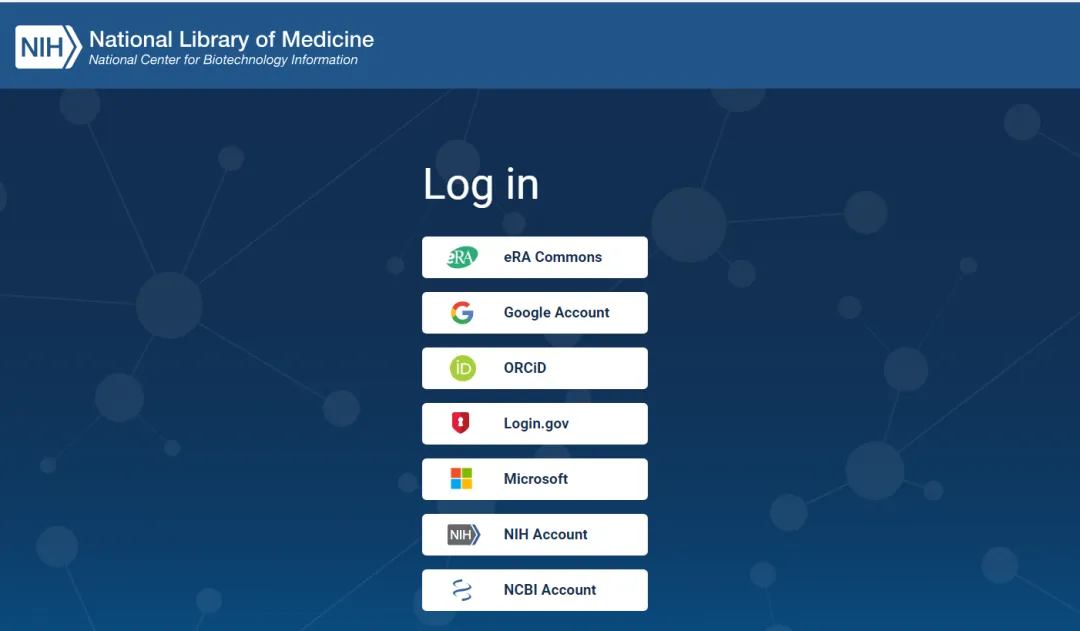
1 注册 NCBI 账号
首先需要登陆https://www.ncbi.nlm.nih.gov/account/注册一个 NCBI 账号,NCBI 支持用各种第三方账户进行注册:
2 准备要上传的原始数据
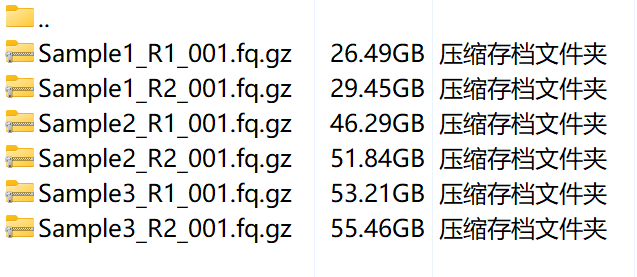
新建一个文件夹,将所有需要上传的样本的原始数据放置到同一个文件中:
3 填写数据信息
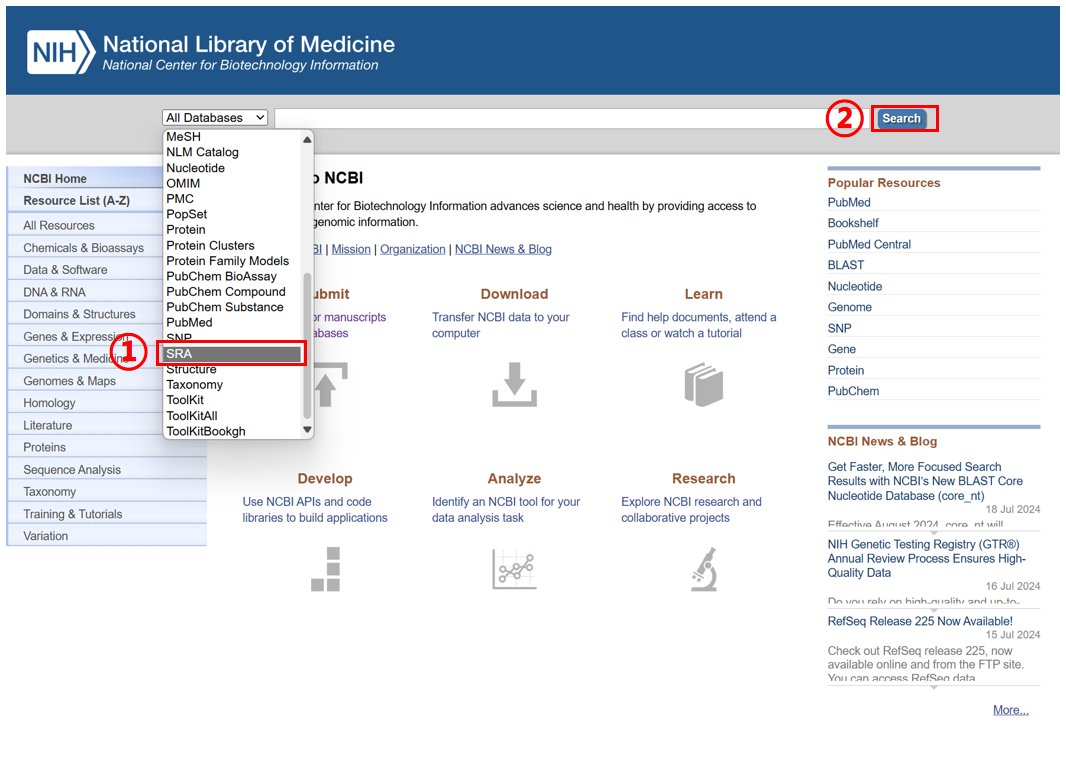
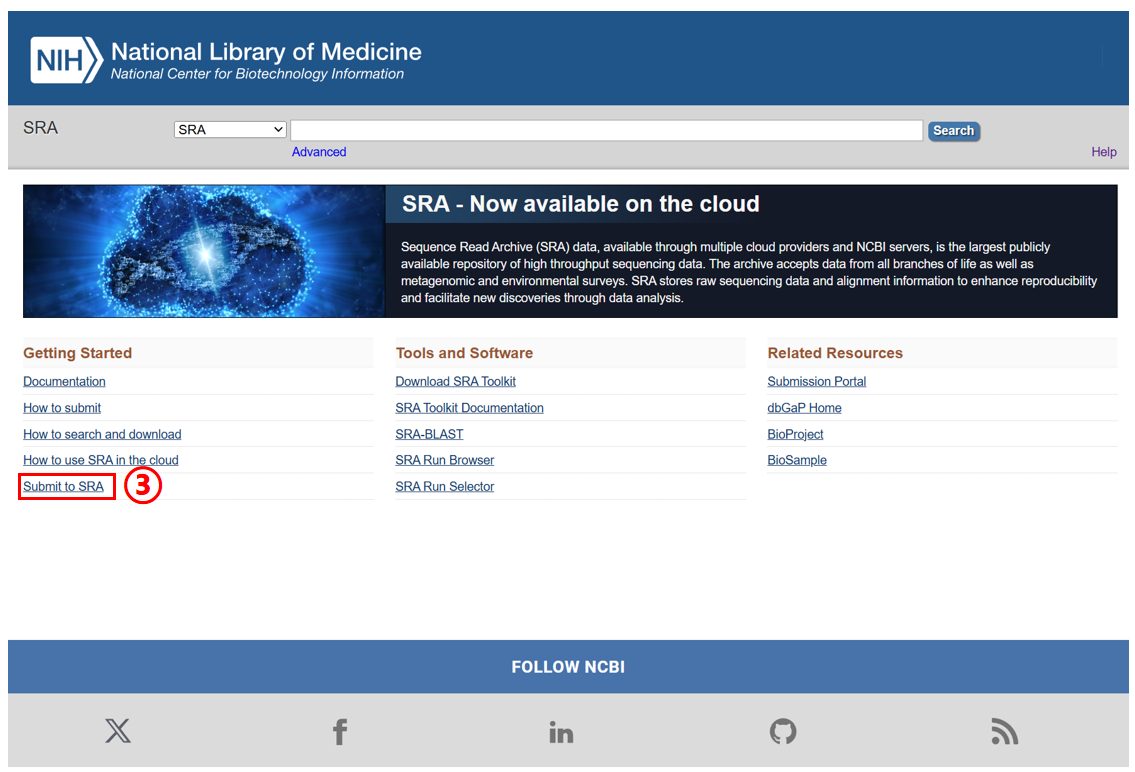
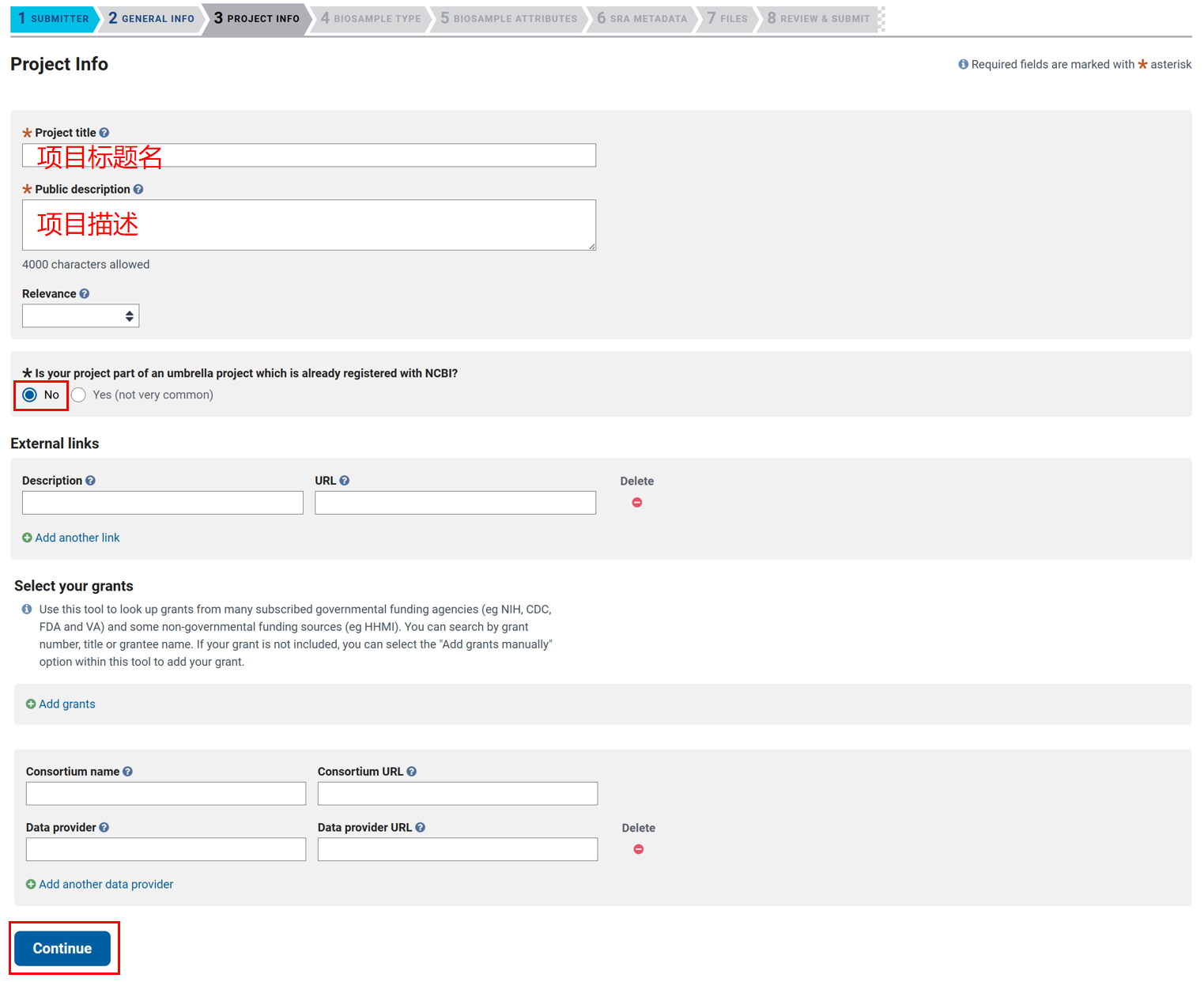
进入 NCBI 首页(https://www.ncbi.nlm.nih.gov/),按如下操作:① 选择 SRA、② 点击 Search、③ 点击 Submit to SRA、④ 点击 New submission
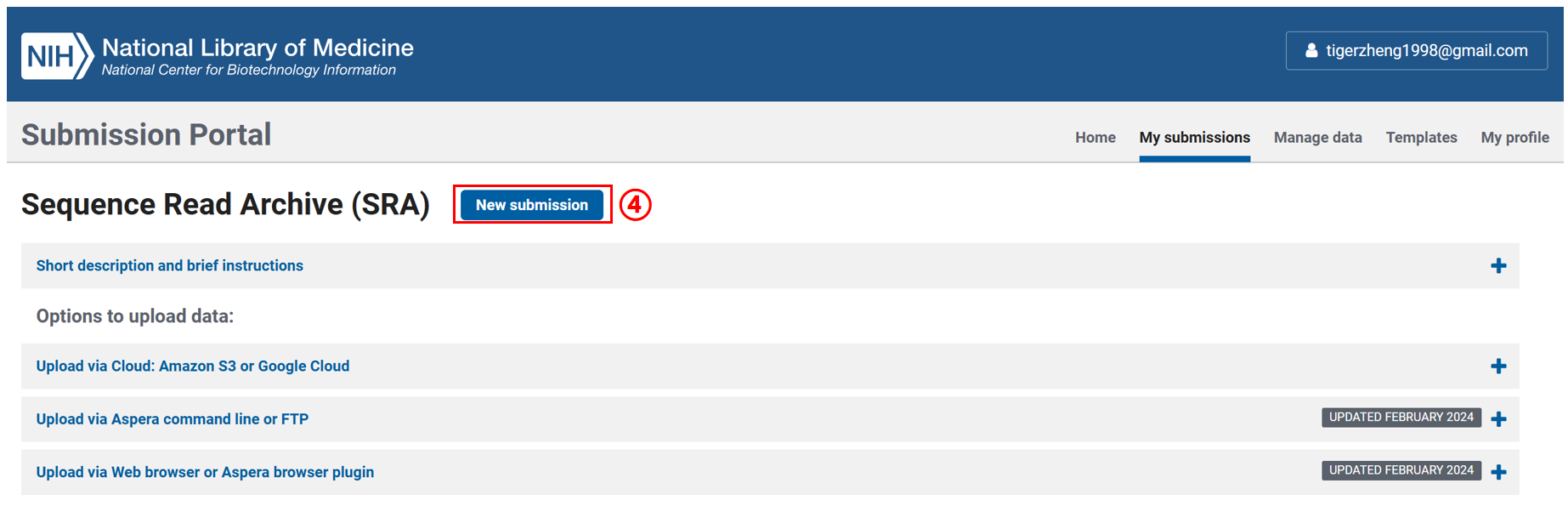
3.1 填写提交者信息(SUBMITTER)
第一项要填写提交者信息,按要求填写即可,注意*为必填选项,填写完成后点击 Continue。
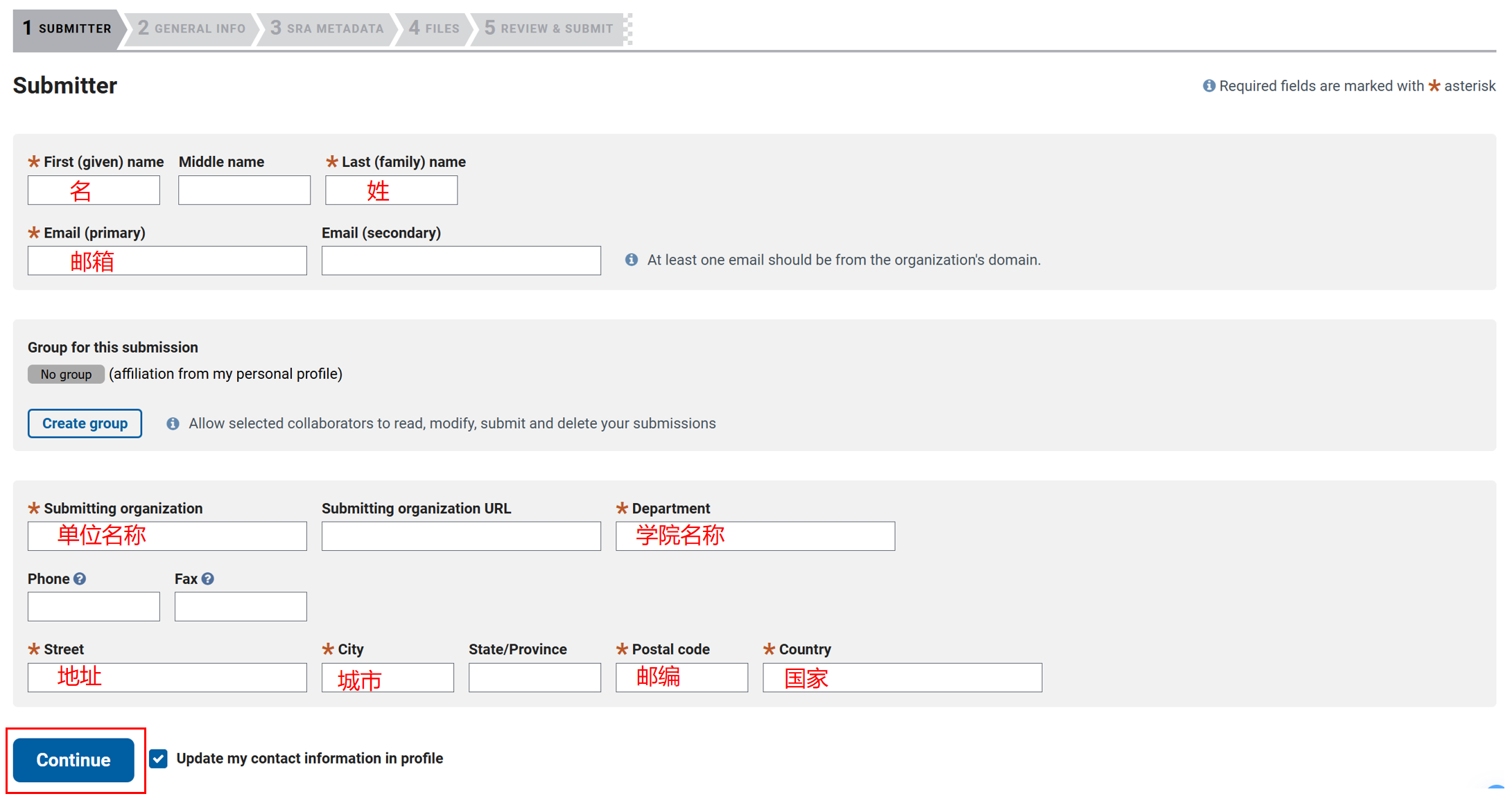
3.2 填写一般信息(GENERAL INFO)
第二项要填写一般信息,如果是第一次提交 BioProject 和 BioSample 都选择 NO,Release data 建议选择靠后一些的日期,避免数据过早发布,后续可以根据课题进展修改。填写完成后点击 Continue。
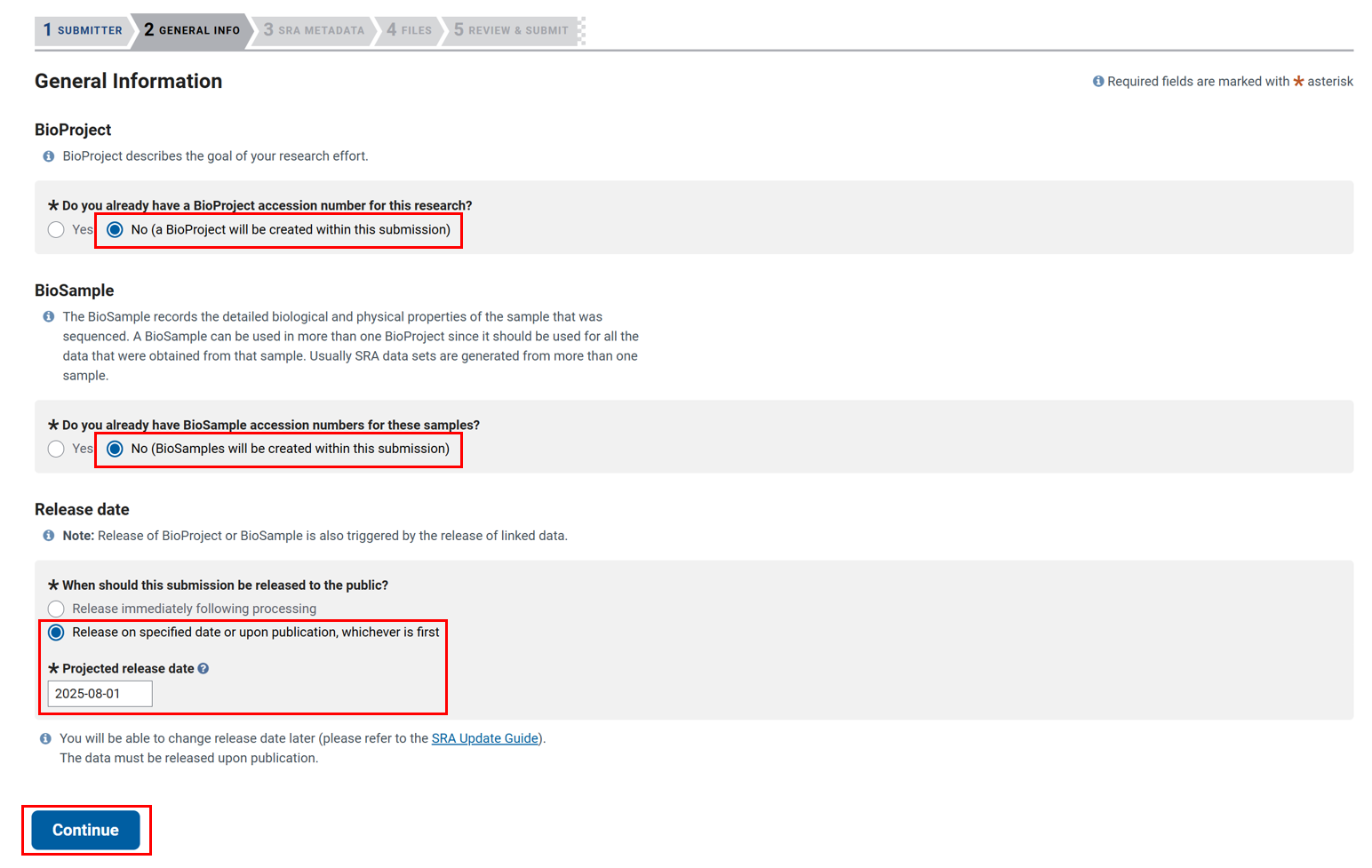
3.3 填写项目信息(PROJECT INFO)
第三项要填写项目信息,填写项目标题、项目描述,如果是第一次提交选择 NO 即可,其他部分选填,填写完成后点击 Continue。
3.4 填写样本类型(BIOSAMPLE TYPE)
第四项要填写样本类型,比如小鼠样本,就选择 Model organism or animal,其他样本在选项中找到对应类型即可,填写完成后点击 Continue。
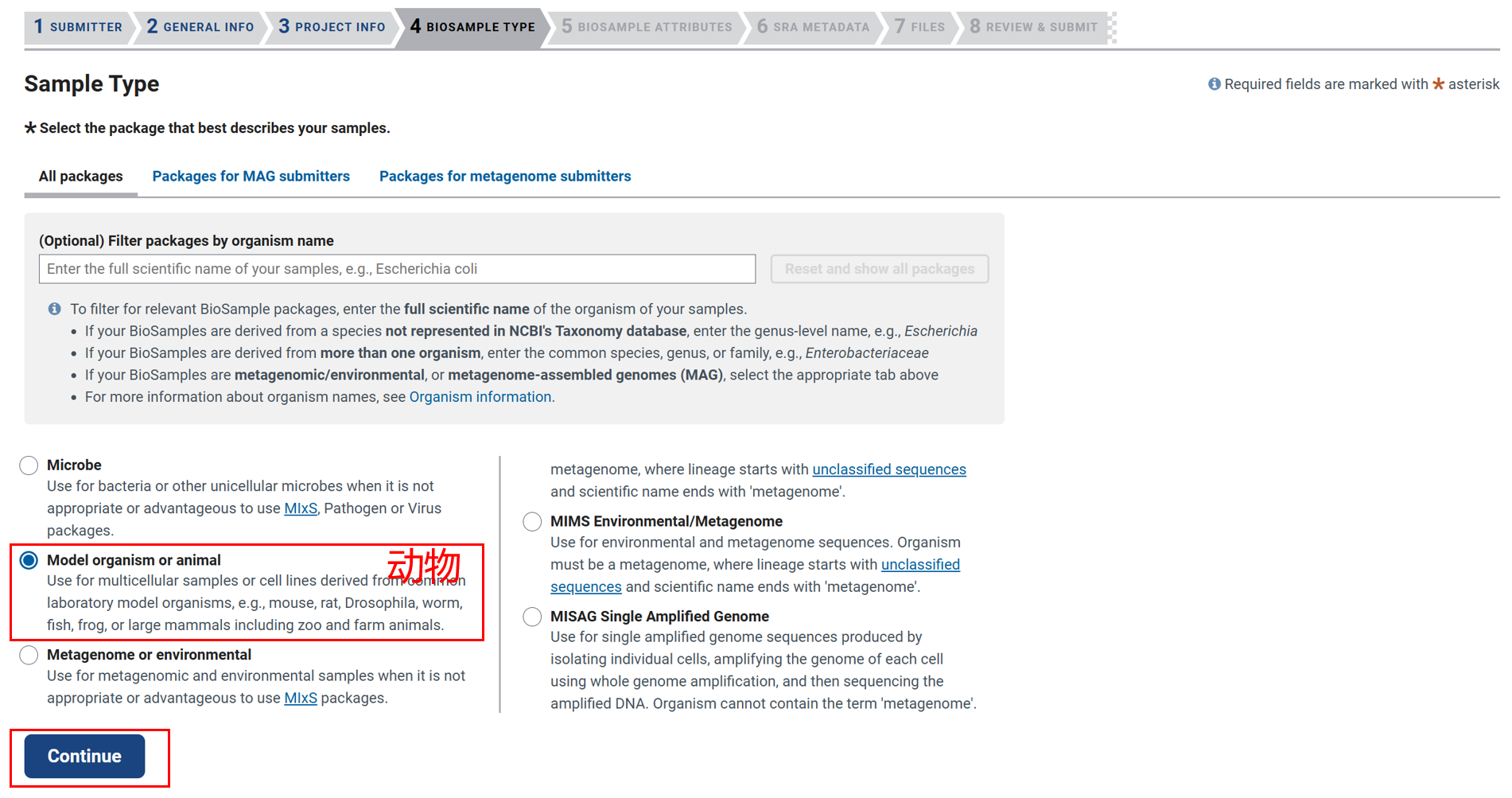
3.5 填写样本属性(BIOSAMPLE ATTRIBUTES)
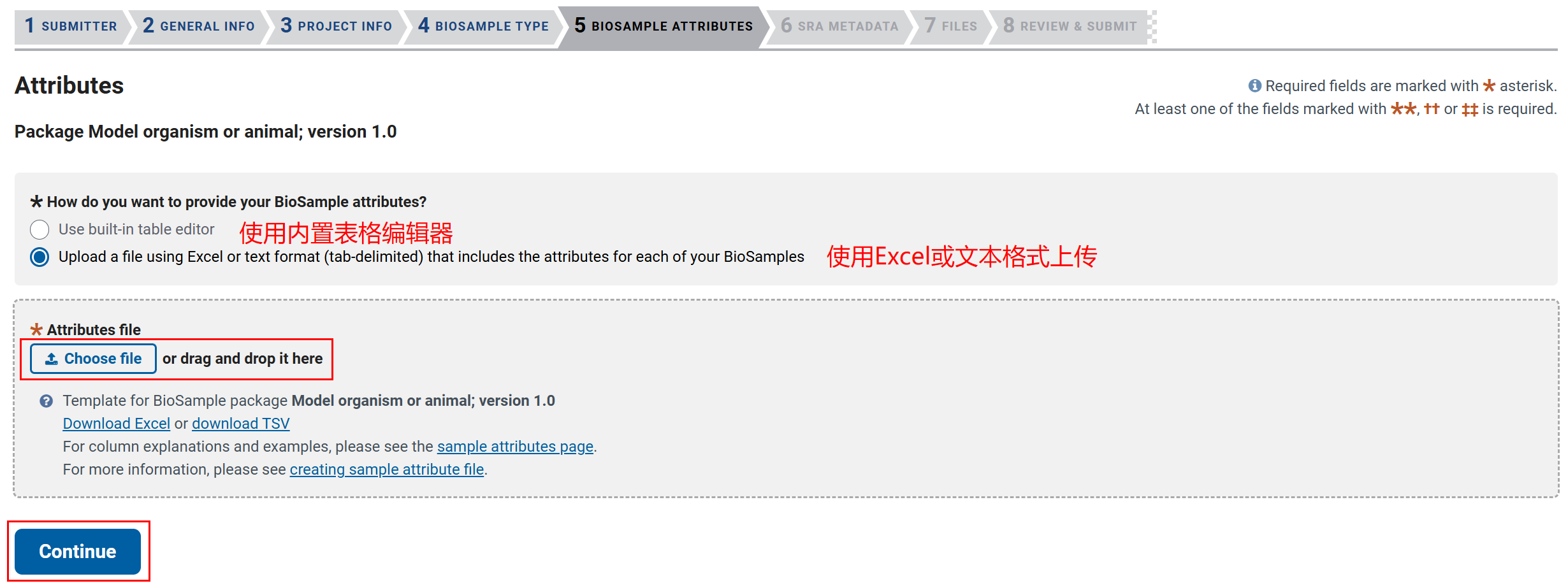
第五项要填写样本属性,可以选择使用内置表格编辑器,或下载Excel和TSV模板填写后上传。
以下载的Excel表格为例:
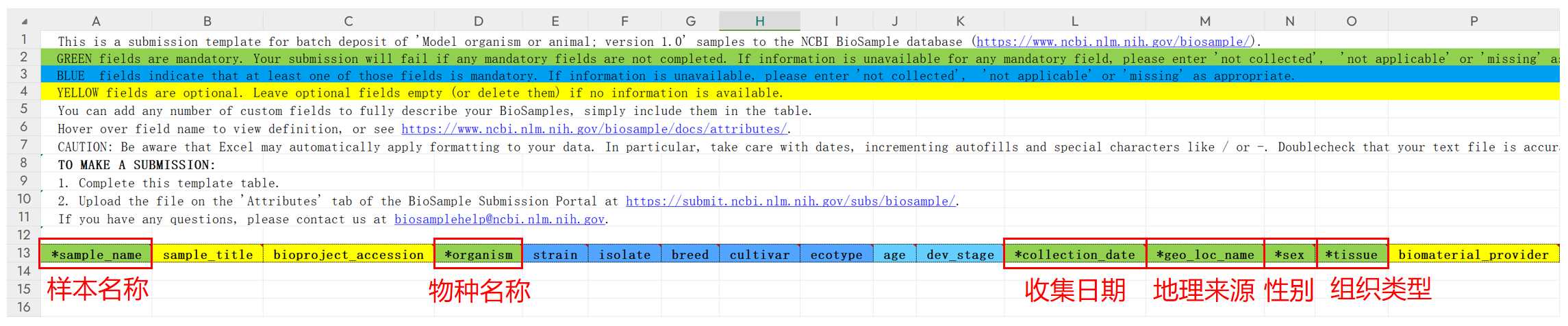
绿色为必填项(必须全部填写),蓝色为选填项(至少选填一个),黄色为可选项(可以空着)。如果任何选项的信息在你的研究中没有涉及,可以填写 "not collected"、"not applicable"、"missing"。你也可以添加任意数量的自定义选项来完整描述你的样本信息。可以将鼠标悬停在选填名称上以查看定义,或者查看 https://www.ncbi.nlm.nih.gov/biosample/docs/attributes/。
填写完成后点击 Choose file 上传,然后点击 Continue。
3.6 填写 SRA Metadata(SRA METADATA)
第六项要填写 SRA Metadata,同样可以选择使用内置表格编辑器,或下载Excel模板填写后上传。
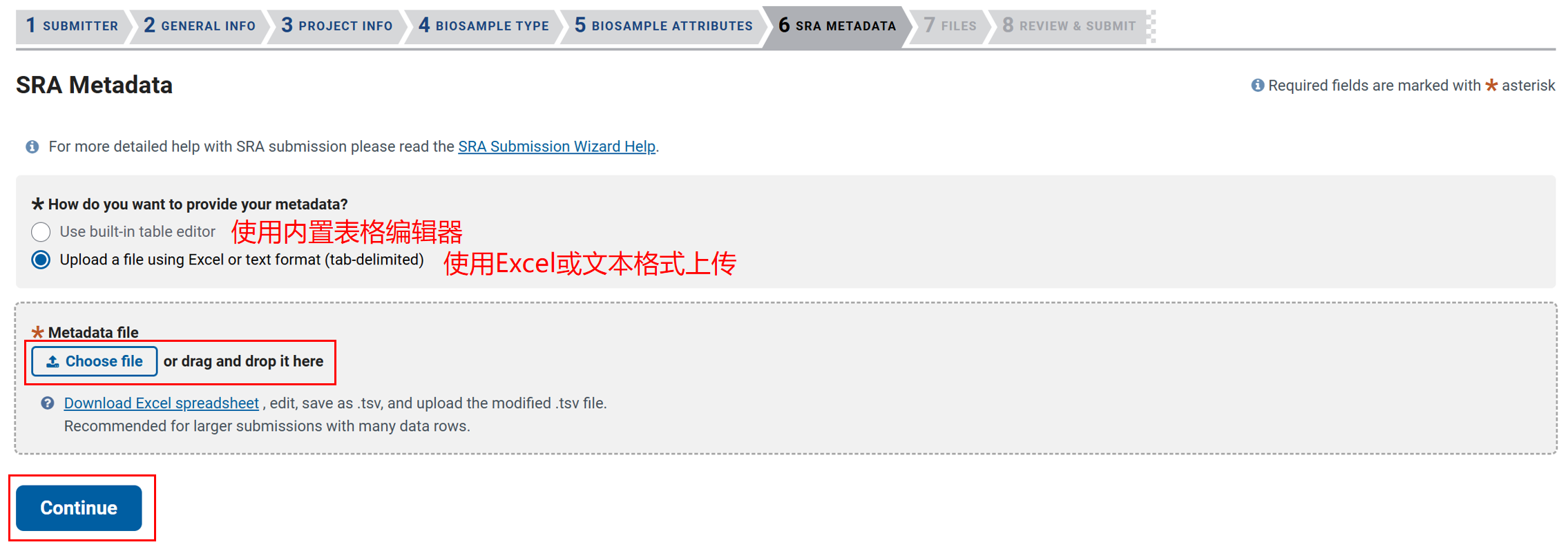
以下载的Excel表格为例:

注意黄色列有下拉菜单,可让从下拉菜单中进行选择。蓝色为必填项,绿色为可选项。每一个选项的填写要求如下:
-
sample_name:样本名称,应该与前一个表格(BIOSAMPLE ATTRIBUTES)中的 sample_name 项名称相同。 -
library_ID:文库ID,必须是唯一的,不能重复。 -
title:数据集的简短描述,格式为 {methodology} of {organism}: isample info},例如 RNA-Seq of mus musculus: adult female spleen。 -
library_strategy:文库策略,如 RNA-Seq。 -
library_source:文库来源,如 GENOMIC。 -
library_selection:文库选择,如 PCR。 -
library_layout:文库设计,single 或 paired。 -
platform:测序平台,如 ILLUMINA。 -
instrument_model:仪器型号,如 Illumina NovaSeq 6000。 -
design_description:设计说明,用于创建测序文库的方法的自由格式描述,简短的"材料和方法"部分。 -
filetype:文件类型,如 fastq。 -
filename:文件名,如 Sample1_R1_001.fq.gz。 -
filename2:文件名2,如双端测序的第二个文件 Sample1_R2_001.fq.gz。 -
assembly:组装,仅当您提交针对 NCBI 组装的 BAM 文件时需要,请提供 NCBI 名称或注册号(例如GRCH37)。 -
fasta_file:fasta 文件,仅当您提交针对 NCBI 组装的 BAM 文件时需要,提供比对过程中使用的自定义组装 fasta 文件的名称(例如 Mouse.fasta)。
填写完成后点击 Choose file 上传,然后点击 Continue。
3.7 上传文件(FILES)
第七项要上传文件。
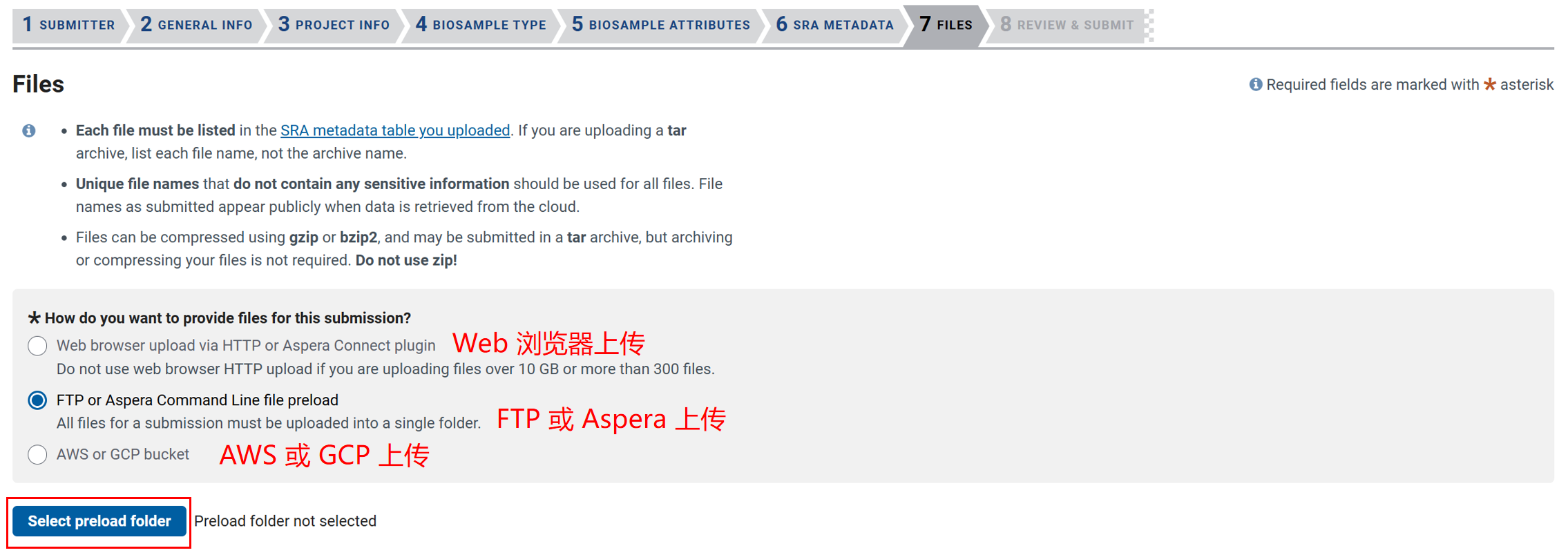
注意事项:
-
上传的每个文件必须在上一步的 SRA metadata 中列出。如果您要上传 tar 存档,请列出每个文件名,而不是存档名称。 -
所有文件都应使用不包含任何敏感信息的唯一文件名,因为文件名会公开显示。 -
文件可以使用 gzip 或 bzip2 进行压缩,并且可以以 tar 存档的形式提交,但不需要存档或压缩文件。 不要使用 zip!
可以通过三种方式上传文件:
-
Web 浏览器上传,通过 HTTP 或 Aspera Connect 插件,但是如果您要上传超过 10 GB 或超过 300 个文件,请勿使用 Web 浏览器 HTTP 上传。 -
FTP 或 Aspera 命令行上传,提交的所有文件必须上传到一个文件夹中。 -
AWS or GCP bucket
由于原始数据通常很大,一般存储在 Linux 服务器中,因此我这里使用 Aspera 命令行上传。Aspera 提供跨越洲际距离的快速上传连接,上传速度可达 100Mb/s。
上传步骤:
-
下载并安装 Aspera Connect 软件,下载链接: https://www.ibm.com/products/aspera/downloads -
下载 key file,下载链接: https://submit.ncbi.nlm.nih.gov/preload/aspera_key/ -
使用以下 Aspera 命令行上传文件: ascp -i <path/to/key_file> -QT -l100m -k1 -d <path/to/folder/containing files> subasp@upload.ncbi.nlm.nih.gov:uploads/tigerzheng1998_gmail.com_M11M5vYX。
其中<path/to/key_file>必须是绝对路径,例如:/home/keys/aspera.openssh。<path/to/folder/containing files>需要指定包含所有要上传的文件的本地文件夹。
上传成功后,点击 Select preload folder 选择上传的文件夹,然后提交。
注意:上传的文件至少需要 10 分钟才能在 Select preload folder 中可供选择。请在创建文件夹后 30 天内完成提交。如果您上传文件但未提交,它们将在文件夹创建后 30 天自动删除。
3.8 检查并提交(REVIEW & SUBMIT)
第八项,检查前面填写的内容,如果没有问题点击 Submit 提交。
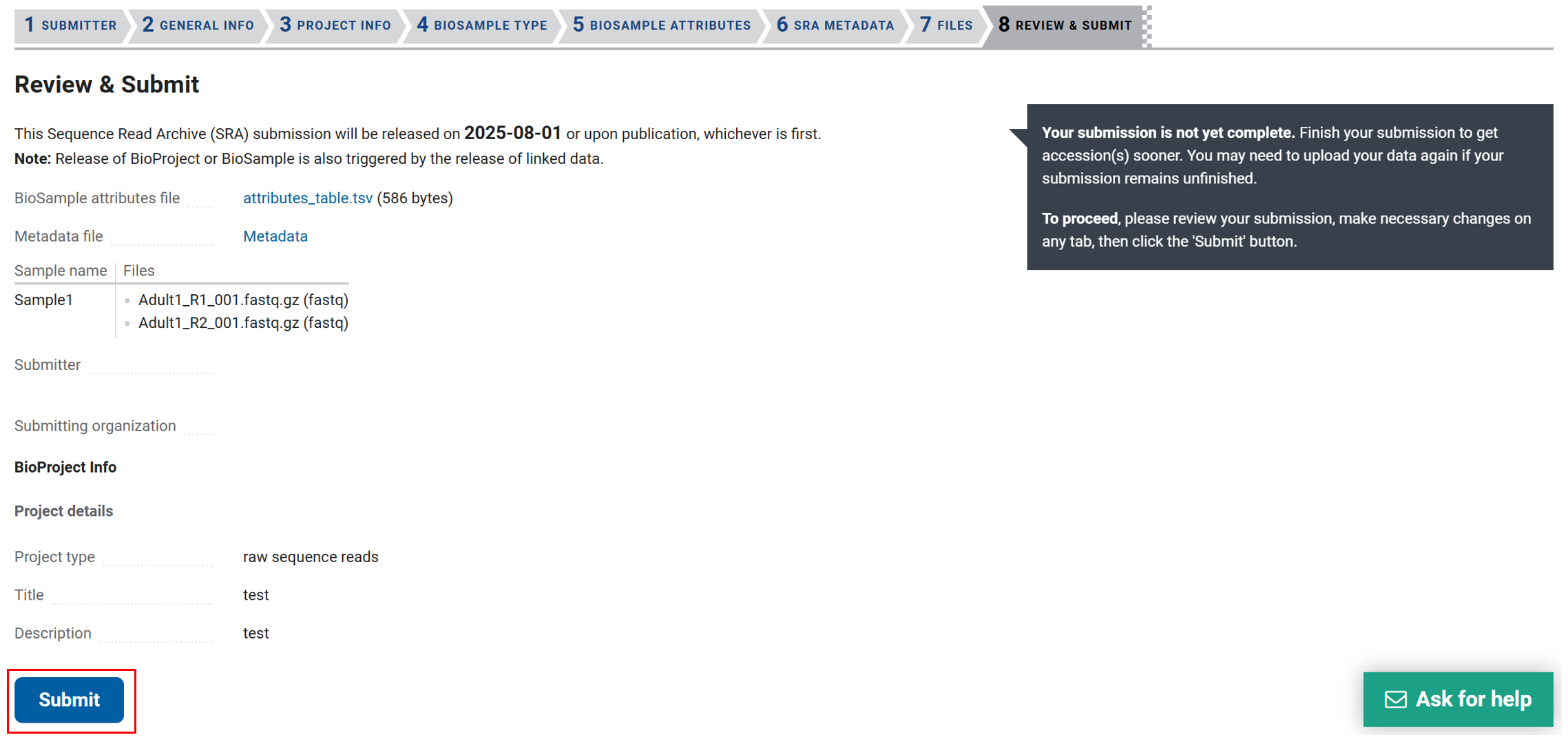
提交后等待 NCBI 审核,SRA 编号可能需要一些时间才能处理完成,一般 24 小时之内能够完成。如果审核长时间没有完成,可以发邮件给 NCBI 询问。如果提交显示下面三项都通过了,就表明数据上传成功了。

注:本文为个人学习笔记,仅供大家参考学习,不得用于任何商业目的。如有侵权,请联系作者删除。

本文由 mdnice 多平台发布
原文地址:https://blog.csdn.net/weixin_45851732/article/details/140638422
免责声明:本站文章内容转载自网络资源,如本站内容侵犯了原著者的合法权益,可联系本站删除。更多内容请关注自学内容网(zxcms.com)!
