差异分析和PPI网路图绘制教程
写在前面
在原文中,作者获得285个DEG,在此推文中共获得601个DEG。小杜的猜想是标准化的水段不同的原因吧,或是其他的原因。此外,惊奇的发现发表医学类的文章在附件中都不提供相关的信息文件,如DEG数据、GO、KEGG富集信息,或是其他相关的文件。唉!!!难道是怕别人复现结果不一致?仅仅提供对读者不关心的文件信息,我们猜想,这是不是期刊要求必须有附件,所以才产生两个文件呢????

获得本期教程数据和代码,后台回复关键词:「20240218」
若我们的分享对你有用,希望您可以「点赞+收藏+转发」,这是对小杜最大的支持。
2.4.1 原文中差异分析

原文中的结果描述,, we screened 471DEGs between therenal fibrosis group and the control group in GSE76882 using the R package “limma”

原文中图形


2.4.2 关于GSE76882数据集
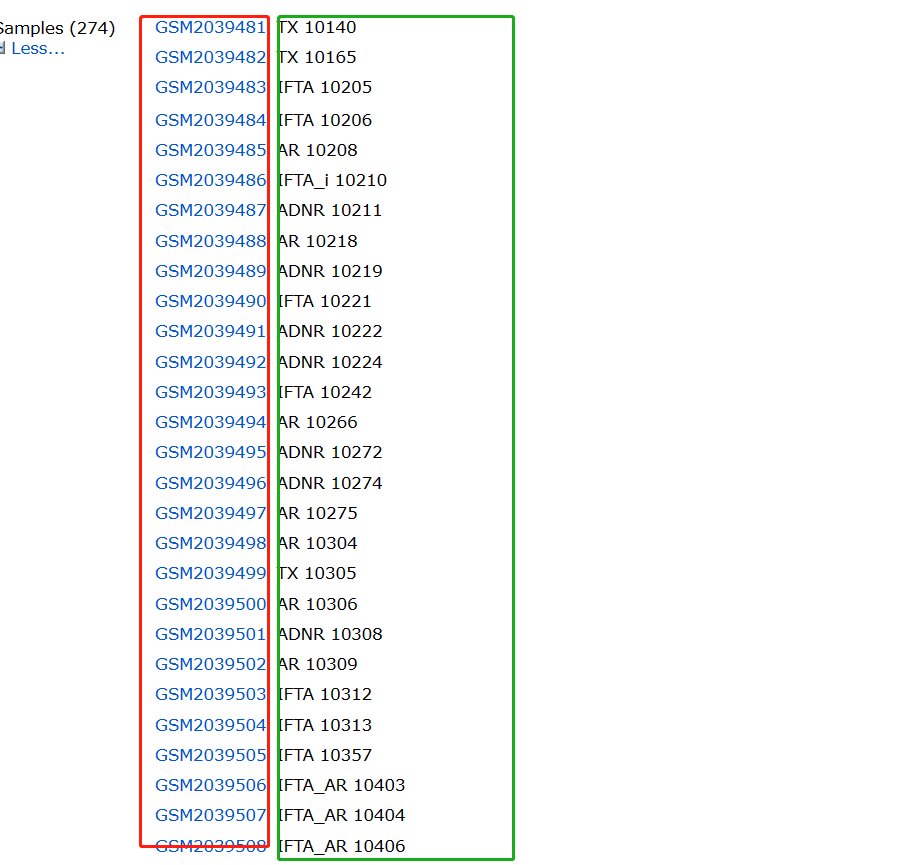
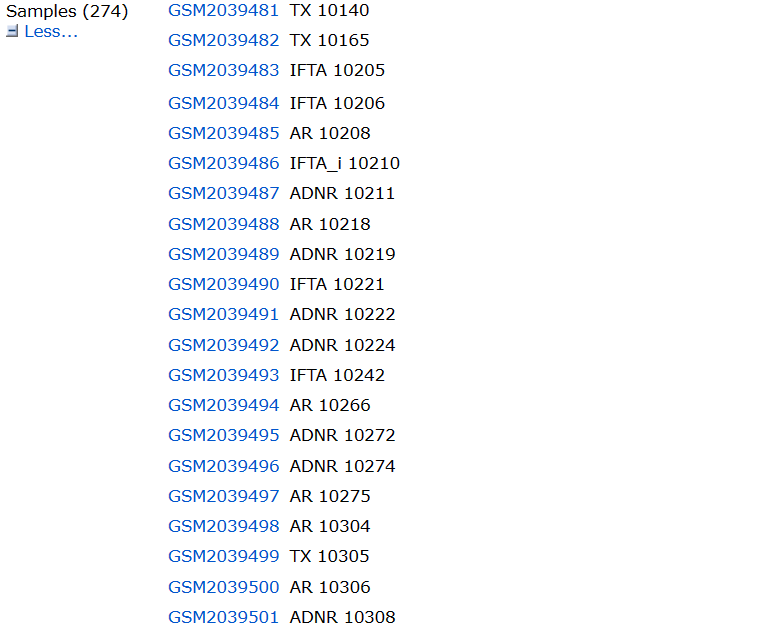
共有274个数据集,其中99个对照组,175个肾纤维化样本。

作者这里就只是简单的分类而已,若细致的分,这里有些数据是可以不被使用的。

对下载的数据集进行分析可获得,前175列数据作为处理组,后99列数据作为对照组。
注意:你需要核对下载后的数据集与GEO数据库中信息是否一致。
2.4.3 差异分析
我们并不知道作者使用那种标准化手段处理数据。首先,我使用log2(x+1)的方式进行标准化,并使用其后面的数据进行差异分析。
2.4.3.1 数据标准化
##'@GSE76882标准化
df02 <- read.csv("00.GEO_RawData/GSE76882_uniq.exp.csv",header = T, row.names = 1)
nor82 <- log2(df02+1)
nor82[1:10,1:10]
write.csv(nor82,"01.GEO_norData/GSE76882_Nor.csv")
2.4.3.2 差异分析代码
-
创建文件夹和导入相关的包
dir.create('02.DEGs_analysis', recursive = TRUE)
library(limma)
library(dplyr)
-
导入数据 csv文件或TXT文件格式
##'##'@读取txt文件格式
#df <- read.table("***.txt", header = T, sep = "\t", row.names = 1, check.names = F)
##'@读取csv文件格式
df <- read.csv("01.GEO_norData/GSE76882_Nor.csv", header = T, check.names = F)
3. 创建比对文件信息 (1) 若你的数据样本不是统一的,需要知道详细信息代表什么。你可以这样创建。
group.list <- c(rep("normal", 25), rep("tumor",24), rep("tumor",42), rep("normal",99)) %>% factor(., levels = c("normal", "tumor"), ordered = F)
获得临床信息方法一
(2)若表达矩阵信息与我们这里一致,那么你可以直接创建即可。
「问:」如何将我们的表达矩阵按分类进行排列。
可以使用下来方法
A. 手动在execl中进行排列,在50个样本数据以内可以使用此方法。
B. 使用一下的方法(仅供参考)
复制这些信息到execl中,排列顺序。

输出样本信息数据

使用R语言进行重新排列矩阵的列
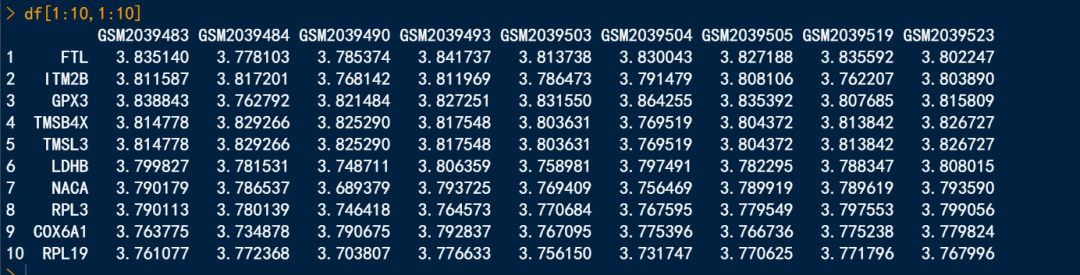
##'@读取csv文件格式
df <- read.csv("01.GEO_norData/GSE76882_Nor.csv", header = T)
df[1:10,1:10]
##'@样本信息顺序,已在execl中排序
df3 <- read.csv("02.DEGs_analysis/001_样本信息.csv",header = F)
head(df3)
##'@样本顺序转换为字符向量
sample_order <- as.character(df3$V1)
##'@对表达量矩阵的列进行重新排列
df_reordered <- df[,c("X",sample_order)]
df_reordered[1:10,1:10]


获取临床信息方法二 「(推荐)」
在下载数据时就需要添加临床信息的参数
2023年《生信知识库》访问网址,此系列专栏已订阅无需重复订阅,订阅后所有教程都可以在此链接中获得。
s
「如下例:」
gset_GSE76882 <- getGEO("GSE76882", destdir = '.',AnnotGPL = T,GSEMatrix =T, getGPL=T)
save(gset_GSE76882 , file = 'GSE76882_eSet.Rdata')
# ## 提取数据
# gset=gset[[1]]
# exprSet1 = exprs(gset)
# #exprSet1 = read.csv("GSE51588.csv",row.names = 1) #####rowname=1很重要
# exprSet1[1:5,1:5]
# # 导出结果
# write.csv(exprSet1, file = "00.GEO_RawData/GSE76882_raw.data.csv",row.names = T,quote = F)
load('GSE76882_eSet.Rdata')
## 提取数据
exp_GSE76882 <- exprs(gset_GSE76882[[1]])
##'依旧推荐使用我们的方法
## 转换ID
##'@加载family.soft文件
anno <-data.table::fread("00.GEO_RawData/GSE76882_family.soft",skip ="ID",header = T)
anno[1:5,1:8]
#colnames(anno)[6] <- "Symbol"
probe2symbol <- anno %>%
dplyr::select("ID","Gene Symbol") %>% dplyr::rename(probeset = "ID",symbol="Gene Symbol") %>%
filter(symbol != "") %>%
tidyr::separate_rows( `symbol`,sep="///")
## 导出 gene symbol数据集合
write.csv(probe2symbol,"00.GEO_RawData/GSE76882_geneSymbol_ID.csv", )
probe2symbol[1:10,1:2]
##
exprSet <- exprSet1 %>% as.data.frame() %>%
rownames_to_column(var="probeset") %>%
#合并的信息
inner_join(probe2symbol,by="probeset") %>%
#去掉多余信息
dplyr::select(-probeset) %>%
#重新排列
dplyr::select(symbol,everything()) %>%
#求出平均数(这边的点号代表上一步产出的数据)
mutate(rowMean =rowMeans(.[grep("GSM", names(.))])) %>%
#去除symbol中的NA
filter(symbol != "NA") %>%
#把表达量的平均值按从大到小排序
arrange(desc(rowMean)) %>%
# symbol留下第一个
distinct(symbol,.keep_all = T) %>%
#反向选择去除rowMean这一列
dplyr::select(-rowMean) %>%
# 列名变成行名
column_to_rownames(var = "symbol")
## 导出数据
write.csv(exprSet,"00.GEO_RawData/GSE76882_uniq.exp.csv",row.names = T)
##----------------------------------------------------------------------------
pd_GSE76882 <- pData(gset_GSE76882[[1]]) # 获取第一个样本的临床信息
group_GSE76882 <- ifelse(str_detect(pd_GSE76882$title, "tumor"), "Tumor", "Normal")
table(group_GSE76882)
group <- factor(group_GSE76882, levels = c("Normal","Tumor"))
## 重新名称
group_list <- ifelse(group == "Tumor", 1,
ifelse(group == "Normal", 0,
NA))
group_list <- as.character(group_list)
limma分析代码
design <- model.matrix(~0 + BC_group, )
colnames(design) <- c("Tumor", "normal")
# Fit a linear model
fit1 <- lmFit(exptotal_df, design)
##
cont.matrix_bc <- makeContrasts(Tumor - normal, levels = design)
fit2 <- contrasts.fit(fit1, cont.matrix_bc)
# Estimate differential expression using eBayes
fit3 <- eBayes(fit2,0.01)
summary(fit3)
#############
tempOutput <- topTable(fit3, coef= 2, adjust.method="BH", sort.by="B", number=Inf)
##
nrDEG = na.omit(tempOutput)
diffsig <- nrDEG
write.csv(diffsig, "01.limmaOut.csv") ## 输出差异分析后的基因数据集
##
## 筛选出差异表达的基因
foldChange = 1
padj = 0.05
All_diffSig <- diffsig[(diffsig$adj.P.Val < padj & (diffsig$logFC>foldChange | diffsig$logFC < (-foldChange))),]
dim(All_diffSig)
write.csv(All_diffSig, "02.diffsig.csv") ##输出差异基因数据集
## 筛选 up and down gene number
diffup <- diffsig[(diffsig$adj.P.Val < padj & (diffsig$logFC > foldChange)),]
write.csv(diffup, "03.diffup.csv")
#
diffdown <- diffsig[(diffsig$adj.P.Val < padj & (diffsig < -foldChange)),]
write.csv(diffdown, "04.diffdown.csv")

2.4.4 绘制火山图
# 绘制火山图
library(ggplot2)
library(ggrepel)
#diffsig <- read.csv("01.TGCA.all.limmaOut-02.csv", header = T, row.names = 1)
data <- diffsig
# 绘制火山图
logFC <- diffsig$logFC
deg.padj <- diffsig$P.Value
data <- data.frame(logFC = logFC, padj = deg.padj)
data$group[(data$padj > 0.05 | data$padj == "NA") | (data$logFC < logFC) & data$logFC > -logFC] <- "Not"
data$group[(data$padj <= 0.05 & data$logFC > 1)] <- "Up"
data$group[(data$padj <= 0.05 & data$logFC < -1)] <- "Down"
x_lim <- max(logFC,-logFC)
###
pdf('02.DEGs_analysis/05.volcano.pdf',width = 7,height = 6.5)
label = subset(diffsig,P.Value <0.05 & abs(logFC) > 1)
label1 = rownames(label)
colnames(diffsig)[1] = 'log2FC'
Significant=ifelse((diffsig$P.Value < 0.05 & abs(diffsig$log2FC)> 1), ifelse(diffsig$log2FC > 1,"Up","Down"), "Not")
ggplot(diffsig, aes(log2FC, -log10(P.Value)))+
geom_point(aes(col=Significant))+
scale_color_manual(values=c("#0072B5","grey","#BC3C28"))+
labs(title = " ")+
## 修改x轴中logFC数值
geom_vline(xintercept=c(-1,1), colour="black", linetype="dashed")+
## 修改Y轴中logP值,基本不会改变,可以忽略
geom_hline(yintercept = -log10(0.05),colour="black", linetype="dashed")+
theme(plot.title = element_text(size = 16, hjust = 0.5, face = "bold"))+
## X/Y轴中命名
labs(x="log2(FoldChange)",y="-log10(Pvalue)")+
theme(axis.text=element_text(size=13),axis.title=element_text(size=13))+
str(diffsig, max.level = c(-1, 1))+theme_bw()
dev.off()

教程复现图

原图
2.4.5 绘制热图
## 绘制差异热图
library(pheatmap)
DEG_id <- read.csv("02.DEGs_analysis/06_DEG_ID.csv", header = T)
## 匹配
DEG_id <- unique(DEG_id$ID)
ID <- as.factor(DEG_id)
head(ID)
dim(ID)
DEG_exp <- df03[ID,]
hmexp <- na.omit(DEG_exp)
#hmexp <- t(hmexp)
hmexp[1:10,1:10]
#write.csv(hmexp, "DEG.Exp.csv")
#
annotation_col <- data.frame(Group = factor(c(rep("normal",99), rep("tumor",175))))
rownames(annotation_col) <- colnames(hmexp)
pdf("02.DEGs_analysis/07.heatmap.pdf", height = 8, width = 12)
pheatmap(hmexp,
annotation_col = annotation_col,
color = colorRampPalette(c("blue","white","red"))(100),
cluster_cols = F,
cluster_rows = F,
show_rownames = F,
show_colnames = F,
scale = "row", ## none, row, column
fontsize = 12,
fontsize_row = 12,
fontsize_col = 6,
border = FALSE)
dev.off()

绘制热图此方法仅是其中一种,大家可以使用前期的教程进行绘制更精美的图形。
2.6.1 PPI网络分析
-
PPI网址 网址:
https://cn.string-db.org/

2. 输入基因ID

3. 选择Organisms,可以选择auto-detect,可以自动识别

4. 点击SEARCH

5. Please wait

6. 点击continue

7. 输出结果

注意:该图形可以进行拖动 8. 可以设置参数,可以默认参数设置

选择超过5个interactors

置信度设置

UPDATE

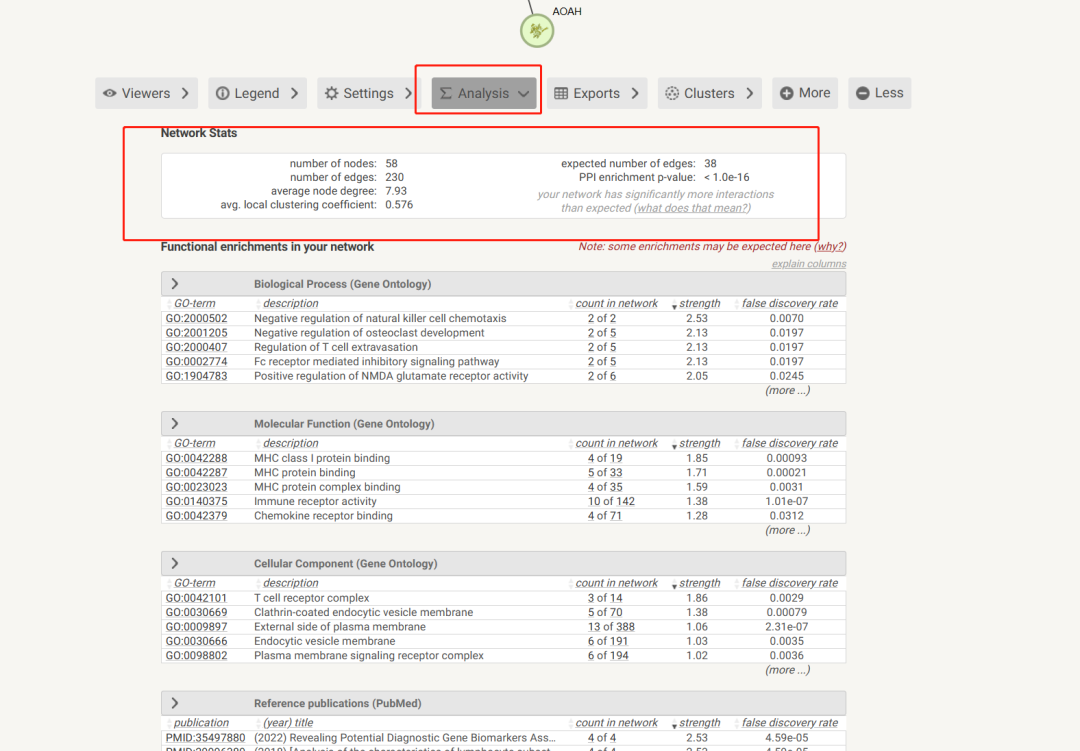
9. Anaysis
10. Exports

2.6.2 下载PPI结果
-
下载图片
-
输出结果文件
-
节点信息

最终分析结果


网络图输入文件

若你的Cytoscape版本较高,可以直接在PPI网页上点击send networkto Cytoscape中,在Cytoscape中直接打开。
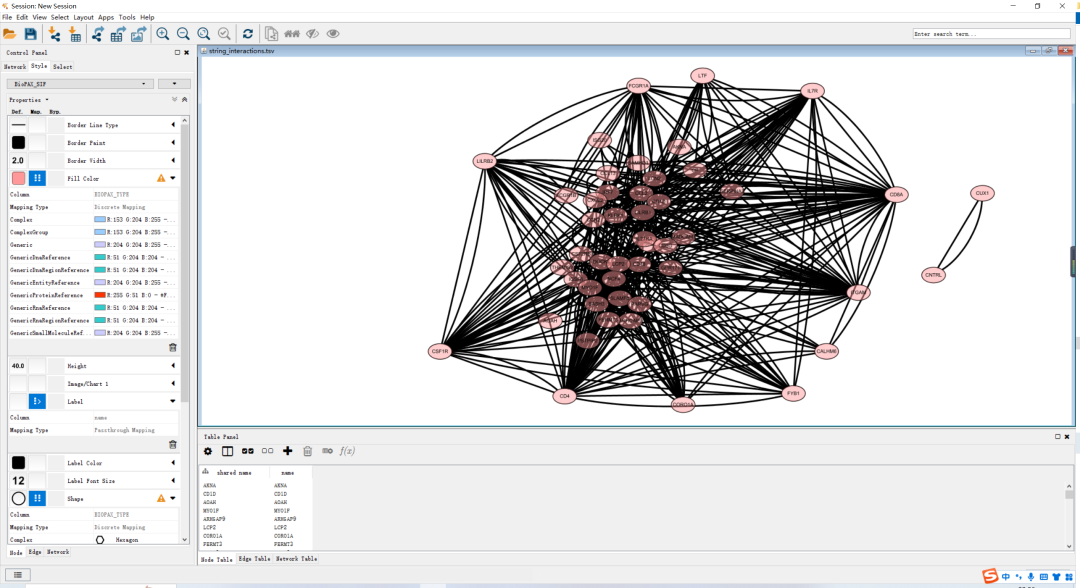
直接使用network节点信息导入,再进行调整即可。
「详细调整参数,可以自己根据网上的教程进行制作即可。」
若我们的分享对你有用,希望您可以「点赞+收藏+转发」,这是对小杜最大的支持。
「往期文章:」
「1. 复现SCI文章系列专栏」
「2. 《生信知识库订阅须知》,同步更新,易于搜索与管理。」
「3. 最全WGCNA教程(替换数据即可出全部结果与图形)」
「4. 精美图形绘制教程」
「5. 转录组分析教程」
「一个转录组上游分析流程 | Hisat2-Stringtie」
「小杜的生信筆記」 ,主要发表或收录生物信息学的教程,以及基于R的分析和可视化(包括数据分析,图形绘制等);分享感兴趣的文献和学习资料!!
原文地址:https://blog.csdn.net/kanghua_du/article/details/136138969
免责声明:本站文章内容转载自网络资源,如本站内容侵犯了原著者的合法权益,可联系本站删除。更多内容请关注自学内容网(zxcms.com)!
